Matem¨¢ticas para descifrar la evoluci¨®n de las especies
La filogen¨¦tica es clave para descubrir la historia evolutiva desconocida

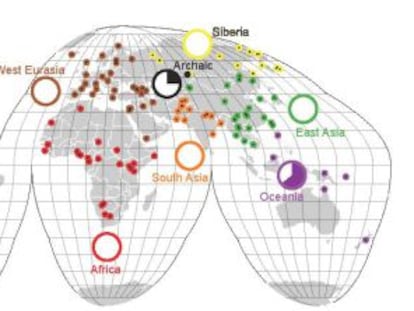
Todas las especies que habitan en el planeta Tierra tienen un antepasado com¨²n. La evoluci¨®n fue dando lugar a diferentes especies, siguiendo caminos a veces misteriosos. Aunque la historia evolutiva de algunos grupos de especies es conocida, quedan por determinar las relaciones de miles de ellas. El genoma es una de las herramientas empleadas para enfrentar este reto, en un campo llamado filogen¨¦tica, en el que colaboran bi¨®logos, estad¨ªsticos, matem¨¢ticos e inform¨¢ticos.
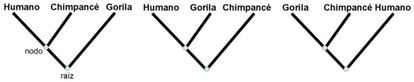
El objetivo final de esta disciplina ser¨ªa poder describir todas las relaciones entre cualquier conjunto concreto de especies, a trav¨¦s de un esquema llamado ¨¢rbol filogen¨¦tico. En este diagrama las hojas representan las especies actuales; la ra¨ªz, el ancestro com¨²n a todas esas especies; los nodos, las especies ancestrales; y la divisi¨®n de ramas, la creaci¨®n de especies. Por ejemplo, al considerar el gorila, el chimpanc¨¦ y el humano, hay s¨®lo tres ¨¢rboles filogen¨¦ticos que podr¨ªan explicar su evoluci¨®n: el que presenta al gorila y al chimpanc¨¦ como especies m¨¢s cercanas (con un ancestro com¨²n), el que une al gorila y humano y el que junta chimpanc¨¦ y humano, como muestra la figura.
A partir del genoma de las especies actuales se intenta deducir qu¨¦ configuraci¨®n, entre todos los posibles ¨¢rboles, parece la m¨¢s apropiada. Como no disponemos del genoma de las especies ancestrales, se emplean modelos de evoluci¨®n de las secuencias de ADN a lo largo del ¨¢rbol. Los par¨¢metros de estos modelos, que desconocemos, son las probabilidades de mutaci¨®n de las secuencias a lo largo del proceso evolutivo en cada rama. A partir de los datos gen¨®micos de las especies actuales, se escoge el ¨¢rbol y los par¨¢metros que maximizan la probabilidad de observar los genomas de las especies actuales. Este proceso de optimizaci¨®n necesita gran cantidad de datos (secuencias de ADN muy largas); como esto no siempre es posible, la inferencia est¨¢ sujeta a error.
Pero, adem¨¢s, este m¨¦todo solo se puede emplear para estudiar grupos de menos de veinte especies, ya que el n¨²mero de ¨¢rboles filogen¨¦ticos va aumentando de forma exponencial seg¨²n el n¨²mero de especies a estudiar. Para tres, s¨®lo hay tres posibles configuraciones, como ya hemos visto. Pero cuando se a?ade una nueva especie, se obtiene un total de quince ¨¢rboles filogen¨¦ticos posibles, y para 50 especies, se obtienen alrededor de 3x1076 posibilidades (este n¨²mero no est¨¢ muy lejos del n¨²mero estimado de part¨ªculas del universo). Este crecimiento de las opciones hace inviable inferir el ¨¢rbol comprobando todos los ¨¢rboles posibles. Una forma de inferir el ¨¢rbol de un grupo grande de especies es usar la aglomeraci¨®n de ¨¢rboles m¨¢s peque?os. Este m¨¦todo tiene sus problemas, ya que las piezas peque?as pueden ser incompatibles entre ellas. Alternativamente, tambi¨¦n se puede construir directamente un ¨¢rbol estimando la distancia evolutiva entre las especies, es decir, estimando la cantidad de mutaciones que han sufrido durante la evoluci¨®n. A partir de aqu¨ª se escoge el ¨¢rbol que minimiza la distancia evolutiva total entre las especies del ¨¢rbol.
Recientemente han surgido t¨¦cnicas que se enmarcan dentro de una nueva disciplina llamada estad¨ªstica algebraica. Estas herramientas est¨¢n basadas en el estudio algebraico de los modelos de evoluci¨®n y permitir¨ªan la inferencia del ¨¢rbol filogen¨¦tico sin tener que estimar las probabilidades de mutaci¨®n del ADN. Estos m¨¦todos est¨¢n en sus primeros pasos de desarrollo, son v¨¢lidos de momento para reconstruir ¨¢rboles con un n¨²mero peque?o de especies y hay que adaptarlos a los m¨¦todos de aglomeraci¨®n para poder construir ¨¢rboles mayores y usarlos con datos reales del genoma. Pero hasta ahora han dado buenos resultados en datos simulados y ya se han implementado en alguno de los paquetes de software m¨¢s usados por los bi¨®logos.
Todos estos esfuerzos en el avance de la filogen¨¦tica no s¨®lo son ¨²tiles para entender la historia evolutiva y, por ejemplo, determinar en qu¨¦ momento se separaron dos grupos de especies, sino que las mismas herramientas se emplean tambi¨¦n en gen¨®mica (para determinar elementos funcionales del genoma), en biomedicina (por ejemplo, para determinar el origen de cepas de virus) e incluso en ling¨¹¨ªstica.
Marta Casanellas es profesora del Departament de Matem¨¤tiques de la Universitat Polit¨¨cnica de Catalunya
Caf¨¦ y Teoremas es una secci¨®n dedicada a las matem¨¢ticas y al entorno en el que se crean, coordinado por el Instituto de Ciencias Matem¨¢ticas (ICMAT), en la que los investigadores y miembros del centro describen los ¨²ltimos avances de esta disciplina, comparten puntos de encuentro entre las matem¨¢ticas y otras expresiones sociales y culturales, y recuerdan a quienes marcaron su desarrollo y supieron transformar caf¨¦ en teoremas. El nombre evoca la definici¨®n del matem¨¢tico h¨²ngaro Alfred R¨¦nyi: ¡°Un matem¨¢tico es una m¨¢quina que transforma caf¨¦ en teoremas¡±.
Tu suscripci¨®n se est¨¢ usando en otro dispositivo
?Quieres a?adir otro usuario a tu suscripci¨®n?
Si contin¨²as leyendo en este dispositivo, no se podr¨¢ leer en el otro.
FlechaTu suscripci¨®n se est¨¢ usando en otro dispositivo y solo puedes acceder a EL PA?S desde un dispositivo a la vez.
Si quieres compartir tu cuenta, cambia tu suscripci¨®n a la modalidad Premium, as¨ª podr¨¢s a?adir otro usuario. Cada uno acceder¨¢ con su propia cuenta de email, lo que os permitir¨¢ personalizar vuestra experiencia en EL PA?S.
?Tienes una suscripci¨®n de empresa? Accede aqu¨ª para contratar m¨¢s cuentas.
En el caso de no saber qui¨¦n est¨¢ usando tu cuenta, te recomendamos cambiar tu contrase?a aqu¨ª.
Si decides continuar compartiendo tu cuenta, este mensaje se mostrar¨¢ en tu dispositivo y en el de la otra persona que est¨¢ usando tu cuenta de forma indefinida, afectando a tu experiencia de lectura. Puedes consultar aqu¨ª los t¨¦rminos y condiciones de la suscripci¨®n digital.